Don't wanna be here? Send us removal request.
Text
Puentes de hidrógeno del ADN
El oxígeno de la molécula del agua va a tener una carga parcial negativa y los hidrógenos van a tener una carga parcial positiva, este acomodo de la molécula le permite hacer un puente de hidrógeno.
Para que haya un puente de hidrógeno, se requiere otro átomo electronegativo de tamaño pequeño, ejemplo el nitrógeno. El nitrógeno todavía tiene un par de electrones libres y tiene una carga parcial negativa, lo que va hacer es una atracción entre el Nitrógeno y el hidrógeno con carga positiva.
A eso se le llama un puente de hidrógeno.
En el ADN forma puentes de hidrógeno,porque hay moléculas dipolares; existiendo nitrógenos que se van a unir a los hidrógenos.
Figura 1. Puentes de hidrógeno ADN; Base pair guanine cytosine (GT) @Yikrazuul

Figura 2. Formación de puente de hidrógeno entre dos moléculas de agua, fuente: @gruporem-ucam

0 notes
Text
AMPK: una vista estructural a través de ChimeraX
María Azucena Jurado Estrada
RESUMEN
Considerando la diversidad de cada proteína dependiendo la combinación de aminoácidos en cada secuencia polipeptídica y sus campos de fuerza que formen, tendrán una estructura única, con un programa extensible para la visualización interactiva y el análisis de estructuras moleculares se van analizar la estructura de la enzima AMPK. Se eligió una proteína de interés usando bases de datos como PBD. A través de determinar distancias, campos de fuerza, aminoácidos se identifican las estructuras supersecundarias Unidades beta-alfa-beta; meandro Beta como parte de las cadenas de la AMPK.
Palabras clave: Aminoácido, estructura 3d proteína, ChimeraX, AMPK, enlaces no covalentes
AMPK: a structural view through ChimeraX
ABSTRACT
Considering the diversity of each protein depending on the combination of amino acids in each polypeptide sequence and their force fields they form, they will have a unique structure, with an extensible program for interactive visualization and analysis of molecular structures. A protein of interest was chosen from databases such as PBD. Through determining distances, force fields, amino acids are identified supersecondary structures beta-alpha-beta units; Beta meander as part of the AMPK chains.
Keywords: Amino acid, 3d protein structure, ChimeraX, AMPK, protein
INTRODUCCIÓN
Las proteínas constituyen la mayor parte de la masa seca de una célula y tienen muchas funciones, un ejemplo de ello: sirven como moléculas de adhesión celular que unen unas células con otras y con la matriz extracelular, como hormonas que transmiten señales desde un grupo de células a otro, como canales iónicos a través de las membranas, así como enzimas (Lieberman & Peet, 2018). Otras proteínas especializadas actúan como anticuerpos, toxinas, fibras elásticas, cuerdas o fuentes de luminiscencia (Alberts, y otros, 2015). Las proteínas son un grupo diverso de macromoléculas, esta diversidad está directamente relacionada con la diversidad de combinación de cada monómero de los 20 aminoácidos que existen en la biología (Mckee & Mckee, 2014); así como su estructura tridimensional. Esto depende de los tres tipos de enlaces no covalentes que participan en el plegamiento de las proteínas (Alberts, y otros, 2015).
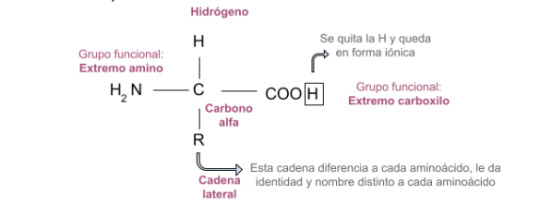
Ilustración 1 Estructura de un aminoácido. Un aminoácido está formado por: (H2N) un extremo amino; (C) un carbono alfa; (CooH) extremo carboxilo (cuando se quita el Hidrógeno, queda en su forma iónica); (H) un hidrógeno y (R) su cadena lateral le da identidad.
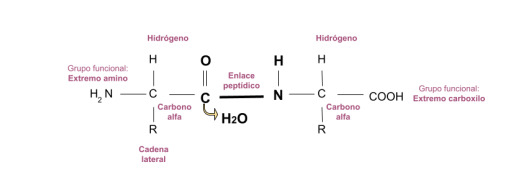
Ilustración 2 Unión de dos aminoácidos. Los aminoácidos se pueden unir entre ellos, se unen de un extremo amino con un extremo carboxilo. Se pueden pegar más aminoácidos y la orientación se mantiene (puede ser de 100, 200 hasta proteínas de 1000 aminoácidos), todas van a tener un extremo amino y un extremo carboxilo.

Ilustración 3 Tipos de enlace que tienen los aminoácidos. Enlace covalente: electrones son atraídos simultáneamente por los dos núcleos atómicos. Un enlace covalente se forma cuando la diferencia entre las electronegatividades de dos átomos es demasiado pequeña para que se produzca una transferencia de electrones para formar iones. Enlaces no covalentes: Estos tres tipos de enlaces no covalentes participan en el plegamiento de las proteínas. Cada uno de estos enlaces por sí solos son débiles, pero muchos de ellos a la vez dan lugar a disposiciones unidas fuertemente. Como resultado de todas estas interacciones, la mayoría de proteínas tienen una estructura tridimensional particular determinada por el orden de aminoácidos de su cadena.
Los científicos distinguen cuatro niveles de organización en la estructura de una proteína, para este artículo nos centraremos en la primera y segunda estructura. En la estructura primaria cada polipéptido tiene una secuencia de aminoácidos específica; por otra parte la estructura secundaria consta de tramos de cadena polipeptídica que forman hélices α y láminas ẞ, estas dos últimas estructuras están estabilizadas por enlaces por puente de hidrógeno entre los grupos carbonilo y grupo amino del esqueleto polipeptídico. Es importante mencionar que los enlaces peptídicos son rígidos, por lo que los carbonos α influyen en los ángulos. (Alberts, y otros, 2015) y (Mckee & Mckee, 2014). La hélice α es una estructura rígida en forma de varilla que se origina cuando una cadena polipeptídica se enrolla en una conformación helicoidal dextrógira. Se forman enlaces por puente de hidrógeno en grupo N-H de cada aminoácido y grupo carbonilo del aminoácido que se encuentra cuatro residuos más adelante. Las láminas plegadas β se forman cuando se alinean dos o más segmentos de la cadena polipeptidica, uno al lado de otro. Cada segmento individual se denomina cadena β. En lugar de estar enrollada, cada cadena β se extiende por completo y se estabiliza por medio de enlaces por puentes de hidrógeno que se forman entre los grupos N-H y carbonilo del esqueleto polipetídico de cadenas adyacentes. Existen dos tipos de estructuras de las láminas β: las paralelas donde los puentes de hidrógeno están dispuestos en la misma dirección y las antiparalelas, los enlaces de puentes de hidrógeno se encuentran en direcciones opuestas; se pueden llegar a observar cadenas paralelas opuestas. Algunas proteínas van a tener combinación de hélices α y lámina plegada β. Estos patrones se denominan estructuras supersecundarias o motivos estructurales. (Mckee & Mckee, 2014).

lustración 4 Estructuras supersecundarias seleccionadas. Unidad α-α; barril β; unidad meandro β; unidades βαβ.
La AMPK (activated protein kinase) en español conocida como proteína cinasa activada. Es una enzima trimérica, constituida por una subunidad α y subunidades β y γ. Es una enzima crucial en la regulación del metabolismo energético celular, cuya actividad se encuentra regulada por diversos mecanismos, entre los cuales el principal es el aumento en la concentración intracelular de AMP. Esta enzima cataliza la fosforilación de múltiples proteínas reguladoras de las vías metabólicas de lípidos y de hidratos de carbono, con lo que favorecen su catabolismo (Hernández-Puga, y otros, 2020). Esta enzima participa en procesos de producción de energía como la glucólisis, la oxidación de lípidos y la gluconeogénesis (Mckee &Mckee, 2014). Considerando la diversidad de cada proteína dependiendo la combinación de aminoácidos en cada secuencia polipeptídica y sus campos de fuerza que formen, tendrán una estructura única, con un programa extensible para la visualización interactiva y el análisis de estructuras moleculares se van analizar la estructura de la enzima AMPK.
MATERIALES Y MÉTODO
Se entró a la base de artículos RCSB y se eligió una proteína de interés, identificando los 4 caracteres propios de la proteína. Se utilizó el programa ChimeraX y con el comando “Open_monbe” espacio de comando y el código previamente identificado, se comenzó a trabajar con la proteína.
Con la opción de control, Action, Color. Se colorea la proteína seleccionada.
En la opción de Tools, Structural Analysys, Distance se calculan dos distancias siempre y cuando se haya seleccionado los átomos que se desea calcular las distancias.
Seleccionando la molécula en la opción de Tools, Structural Analysys, H-Bonds se marcan los puentes de hidrógeno.
En la opción de Tools, Structural Analysys, angles/torsion se marcan los ángulos, siempre que se selecciones tres átomos.
En la opción de Tools, Structural Analysys, match maker se pueden contraponer dos proteínas.
La reconstrucción de proteínas en Swiss Model, es necesario que estén en formato fasta y de ahí convertirlos en código ordenado con números.
Una vez que Swiss Model reconstruye proteínas, es importante que sí aparecen dos modelos, guiarse por la barrita azul de coverage, por lo general el modelo 1 es el que baraca más y se parece más en identidad, la barra coverage indica en cuanto porcentaje se reconstruyó. Así como el QMEANDisCo Global, indica la calidad de la proteína que va de 0.5 a 1. Siempre muestra dos colores, naranja y morado; el morado representa mayor calidad de la proteína.
RESULTADOS
Práctica 1: Elegir una proteína e irse adaptando al programa

Figura 1. Seleccionar proteína de interés. Con ctl se elegía un aminoácido.
Práctica 2: marcar segunda estructura B plegada y colorear las cadenas de distintos colores.
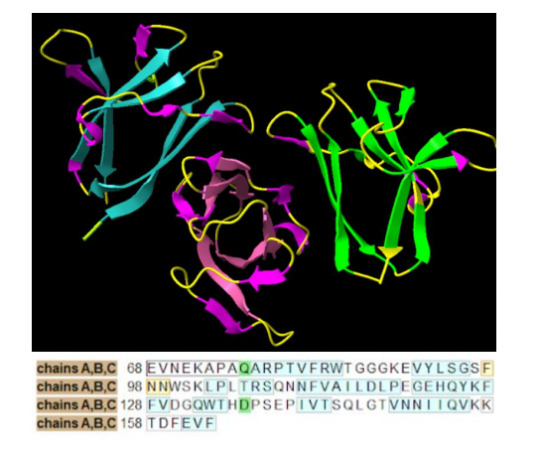
Figura 2. Seleccionar segunda estructura B e identificar las cadenas de la proteína
Práctica 3: identificación de aminoácidos.

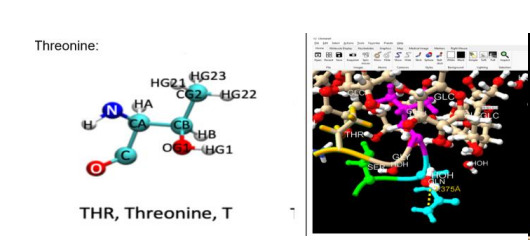
Figura 3. Identificación de aminoácidos en la proteína
Práctica 4: Calcular distancias

Figura 4: Calculo de distancias
Práctica 5: Calcular de fuerzas de Van Der Waals y puentes de hidrógeno
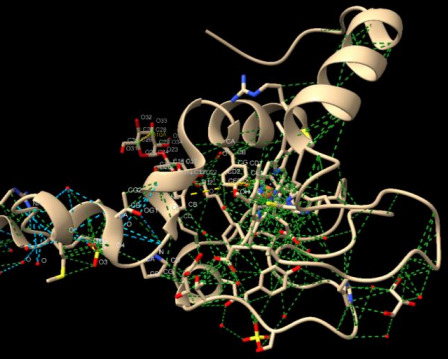
Figura 5. Fuerzas de Van Der Waals y puentes de hidrógeno
Práctica 6: Calcular ángulos

Figura 6. Cálculo de ángulos
Práctica 7: Contraponer dos modelos de la misma proteína pero de diferente especie

CONCLUSIÓN Se documentó la estructura de una proteína, aminoácido y enlaces, se ubicó la práctica teórica a partir de la visualización interactiva y se realizó el análisis de estructuras moleculares en programas como ChimeraX y Swiss Model. A través de determinar distancias, campos de fuerza, aminoácidos se identifican las estructuras supersecundarias Unidades beta-alfa-beta; meandro Beta como parte de las cadenas de la AMPK. Se identificaron ciertas variaciones en los loops de la AMPK de origen humano y mosca. Así como en la revisón mutaciones del AMPK en humanos. Lo visto en teoría coincide con el modelado de proteínas.
REFERENCIAS BIBLIOGRÁFICAS
Mckee, T., & Mckee, J. R. (2014). Bioquímica Las bases moleculares de la vida. En T. Mckee, & J. R. Mckee, Bioquímica Las bases moleculares de la vida (págs. 110-141, 406- 408). México: McGRAW- HILL.
Alberts, B., Johnson, A., Lewis, J., morgan, d., Raff, M., Roberts, K., & Walter, P. (2015).Biología Molecular de LA CÉLULA Sexta Edición. En B. Alberts, A. Johnson, J. Lewis, d. morgan, M. Raff, K. Roberts, & P. Walter,Biología Molecular de LA CÉLULA extaEdición (págs. 109-135). Estados Unidos de América: Garland Science.
Lieberman, M., & Peet, A. (2018). Marks Bioquímica médica básica. En M. Lieberman, & A. Peet, Marks Bioquímica médica básica (págs. 80-86). Philadelphia: Wolters Kluwer.
Hernández-Puga, G., Laguna-Maldonado, K. D., Gregg-García, R., Barrera-Zárate, G., LópezOrtiz, F. D., & Matuz-Mares, D. (2020). La AMPK como diana terapéutica del síndrome metabólico. Revista Médica del Instituto Mexicano del Seguro Social, 612- 621.
ANEXO:
Elegir una proteína e irse adaptando al programa.

Figura Anexo 4.1 Seleccionar proteína de interés.
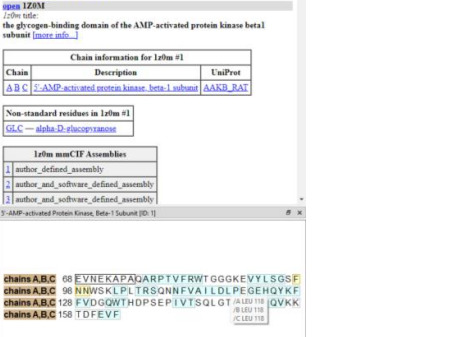
Figura Anexo 4.2 Ver secuencia de los aminoácidos
Práctica 2: marcar segunda estructura (beta plegada)
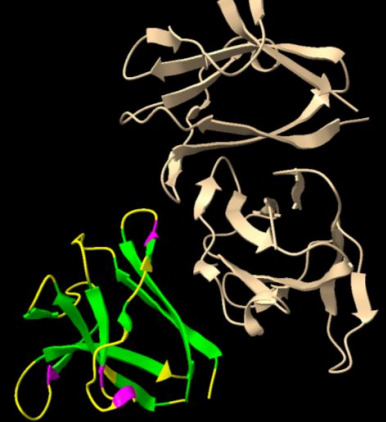
Figura Anexo 4.3 marcar segunda estructura (beta plegada)
Identificar CADENA A

Figura Anexo 4.4 Identificar cadena A
Identificar CADENA b

Figura Anexo 4.5 Identificar cadena B
Identificar CADENA c

Figura Anexo 4.6 Identificar cadena C Encontrar patrón de aminoácidos por cada cadena
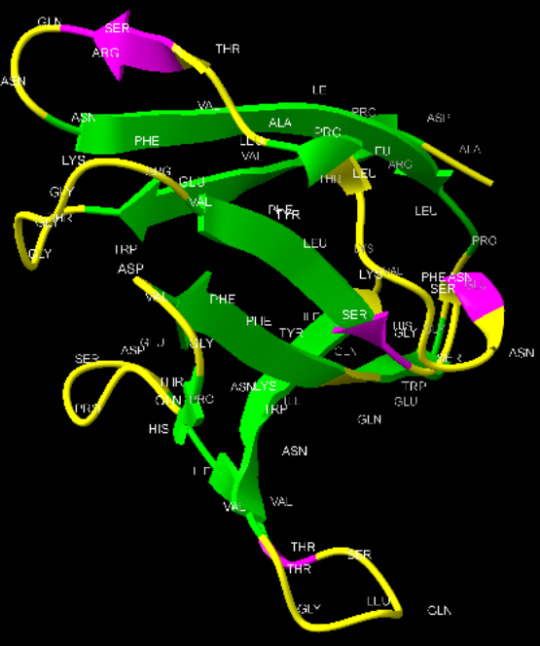
Figura Anexo 4.7 Encontrar patrón de aminoácidos por cada cadena C
BUSCAR MUTACIONES
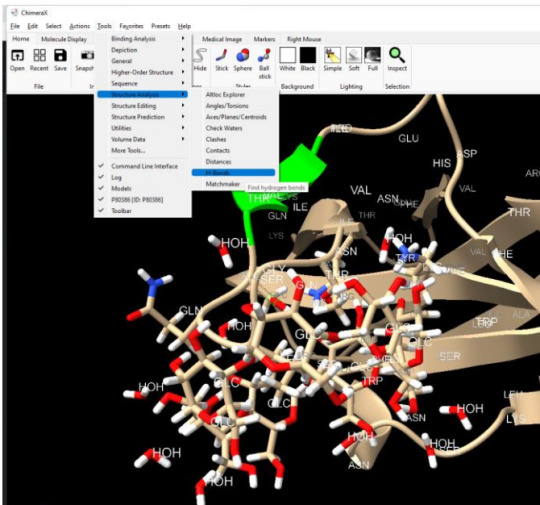
Figura Anexo 4.8 Buscar mutaciones Serina carbono Beta
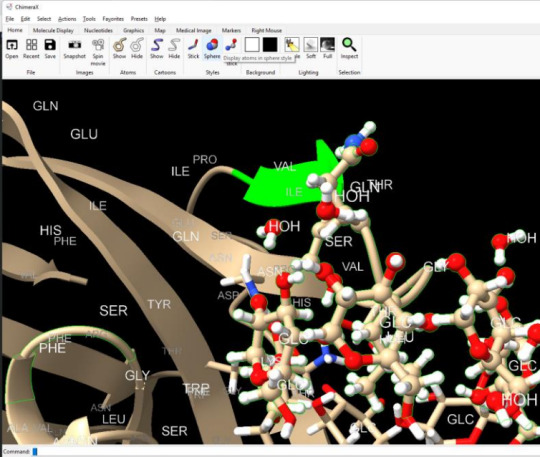
Figura Anexo 4.9 Buscar mutaciones Proline.
Práctica 3: identificación de aminoácidos en la proteína seleccionada.
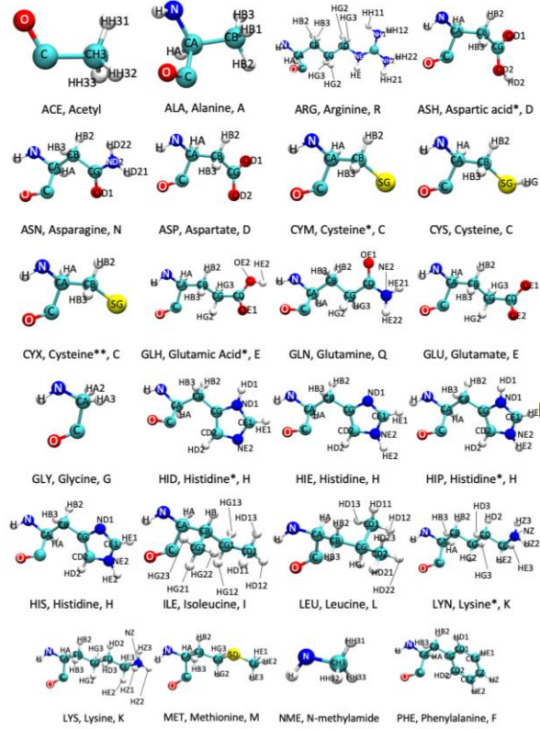
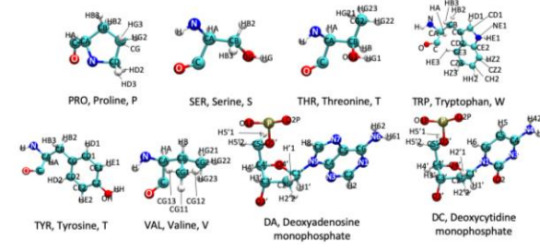
Figura Anexo 4.10 diagrama de estructuras usada para la identificación de aminoácidos
4. Cálculo de distancias

Figura Anexo 4.11 Tools / secuence analysis/ distance
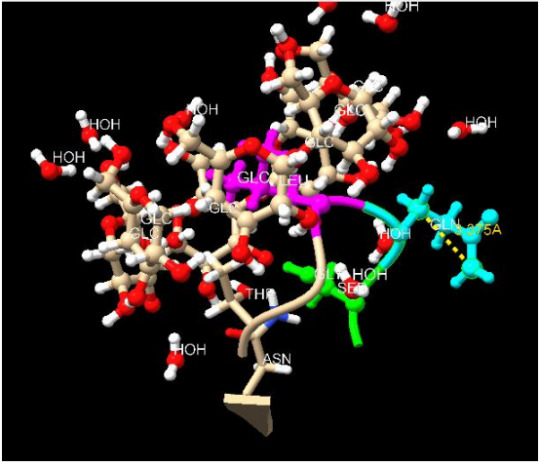
Figura Anexo 4.12 Calculo de distancias
5. Calcular de fuerzas de Van der Waals y puentes de hidrógeno


Figura Anexo 4.13 fuerzas de Van der Waals y puentes de hidrógeno
6. Calcular ángulos


Figura Anexo 4.14 Calcular ángulos
Contraponer dos modelos de la misma proteína pero de diferente especie. Proteínas de interés: seleccionar la misma proteína con la que se está trabajando en diferentes especies
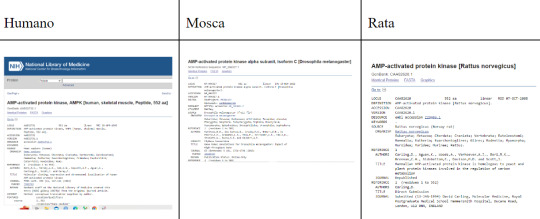
Figura Anexo 4.15 Buscar misma proteína diferentes especies.
1 note
·
View note
Text
Construyendo teóricamente mi primer biorreactor (ciclo del azufre)
Introducción
La mayor parte del azufre de nuestro planeta, se encuentra en sedimentos y rocas en forma de minerales de sulfato, principalmente como yeso ( Ca SO4 ) y minerales de sulfuro (pirita,Fe S2), aunque el mar constituye el principal reservorio de sulfato de la biósfera. Una parte significativa de azufre particularmente el dióxido de azufre (SO2 gas ), se incorpora al ciclo de azufre mediante la actividad humana, sobre todo por combustión de combustibles fósiles.
El sulfuro de hidrógeno (HS- )o ( H2S ) es el principal gas volátil del azufre. Esta sustancia se forma principalmente por reducción bacteriana de sulfato
(SO42- +4H2 H2S +2 H2O + 2OH- ) o por las emisiones geoquímicas o por las emisiones geoquímicas de fuentes de sulfuro y volcanes. Aunque el( H2S ) es volátil, la forma de sulfuro presente en el ambiente depende del pH: por debajo de valores de pH 7 predomina el ( H2S ) y por encima de 7 predominan las formas no volátiles el ( SH-) y el (S2- ). En conjunto el ( H2S ), ( SH-) y el (S2- ) se denominan como sulfuros. El ( H2S ) es tóxico para muchas plantas y animales, cuando se combina con el hierro de los citocromos, bloquea la respiración, por lo cual el sulfuro es considerado tóxico. Su formación usualmente es a partir de la reducción de sulfato. La detoxificación en el ambiente del ( H2S ) se lleva a cabo mediante la reacción con compuestos de hierro, para formar (Fe S )pirrotina y ( FeS2 )pirita. El color negro de muchos sedimentos sulfúricos o de los cultivos de bacterias reductoras de sulfato se debe a estos minerales de sulfuros metálicos donde se produce sulfato reducción se debe a la acumulación de FeS.
Se realizó una búsqueda en el manual de Bergeys y se identificó a dos posibles cepas idóneas para la oxidación de sulfuro a sulfatos y al mismo tiempo se disminuya el PH.
Objetivo
Sabiendo que el (HS-) o ( H2S ) es tóxicos para muchas plantas y animales se Identificará una cepa bacteriana, que ayude a estabilizarlos bioquímicamente transformándolos en (SO42-), así como elevación y se puedan incorporar al suelo de los cultivos haciéndolos más productivos.
Método
Se encontraron dos cepas bacterianas, que ayudan a transformar a través de la oxidación el (SO42-) a ( H2S ), así como elevación de su PH.

Bioquímica (metabolismo del azufre)

Revisión taxonómica
Especies elegidas para construcción de biorreactor:



Método para elaborar un biorreactor que transforme de ( H2S ) a (SO42-) y eleve los niveles de PH
Se seleccionan dos bacterias Thiosphaera pantorropha y Thiobacillus ferrooxidans capaces de oxidar el sulfuro y transformarlo a sulfato, así como elevar PH.
Se aíslan 6 tubos bacteriológicos de cada cepa acorde a su medio de cultivo, en el caso de las Thiobacillus ferrooxidans se elige un medio de cultivo acuoso rico en arsénico y azufre para su crecimiento. Para la Thiosphaera pantorropha se usa un agua rica en azufre.
Se deja incubar a temperatura ambiente.
Posterior se elige un medio de cultivo:
Agar Xilosa Lisina Desoxicolato (XLD), revelan la formación de ácido sulfhídrico (H2S) por la precipitación de sulfuro de férrico (Fe2S3) de color negro presente en las colonias; el viraje es de color rojo púrpura alrededor de las colonias cuando existe un aumento de PH
Con las muestras de las cajas de Petri que cambiaron su viraje y dieron positivas a lo buscado, se aplica tinción gram y se hace una tabla de caracterización.

Identificar morfotipos (rasgos genotípicos para decidir qué línea se escoge de progenitores para hacer la conjugación).
Se deja incubar a temperatura ambiente y nuevamente se inoculan en los medios de cultivo previamente seleccionados.
Se hace una tabla de caracterización y se eligen las cepas que se van a poner a prueba.
Planificación de la prueba reto, donde se añada la bacteria a un medio controlado acuoso donde normalmente existen emisiones altas de ( H2S ) y un PH bajo.
Cuantificación de ( H2S ) y (SO42-) antes y después de comenzar la prueba.
Teniendo el (SO42-) que se utiliza para la elaboración de biofertilizantes , se ocupan en el suelo de los cultivos ayudando a su productividad.
Conclusión
Se encontraron dos cepas bacterianas, que ayudan a transformar a través de la oxidación el (SO42-) a ( H2S ), así como elevación de su PH.
Discusión de resultados
Se documentaron varios medios de cultivo, donde se pudiera medir la oxidación de ( H2S ) y que se transformara a (SO42-), sin embargo el más cercano que nos puede servir para este biorreactor es el Agar Xilosa Lisina Desoxicolato (XLD), aunque no es específica se eligió por que su viraje indica cuando elementos del azúfre bajan el PH cambiando su viraje de negro a rojo morado. Así mismo en la elección de cepas, se documentó de la bacteria paracoccus pantotrophus, que oxida de sulfuro y tiosulfuro y son reducidos a sulfato de azufre y este está formado por más de quince genes que codifican varios citocromos y otras proteínas necesarias para la oxidación directamente a sulfato de compuestos de azufre. Sin embargo a pesar de ser idónea, no se consideró ya que no se encontraba en el manual de Bergey´s.
Referencias
Chandra, T. S., & Friedrich, C. G. (1986). Tn5-induced mutations affecting sulfur-oxidizing ability (Sox) of Thiosphaera pantotropha. j bacteriol, 446–452.
de La Guarda, M. R. H. G. (2021). Análisis genómico de plásmidos de Acidithiobacillus ferrivorans PQ510 y Acidithiobacillus ferrooxidans PQ506 aislados de una zona minera de Cerro de Pasco. Universidad Nacional Mayor de San Marcos.
de Microbiología, I. V. (2012). Bacterias productoras de H2S: importancia en el deterioro de productos pesqueros y cárnicos - Cultivo cualitativo y cuantitativo; identificación molecular (PCR y Secuenciación). Instituto Valenciano de Microbiología.
García de la Guarda, R. H. (2021). Análisis genómico de plásmidos de Acidithiobacillus ferrivorans PQ510 y Acidithiobacillus ferrooxidans PQ506 aislados de una zona minera de Cerro de Pasco - Perú.
HACH. (2023). Prueba BART para bacterias reductoras de sulfato.
Holt, J. G., Krieg, N. R., Sneath, P. H. A., Staley, J. T., & Williams, S. T. (2000). Bergey´s Manual of DETERMINATIVE BACTERIOLOGY (Ninth Edition). Lipincoott wiliams & wilkins.
Lozano, L. C., Ramírez, L. C. C., & Ramírez, L. C. C. (2021). Las bacterias, su nutrición y crecimiento: una mirada desde la química. Nova, 19(36). https://doi.org/10.22490/24629448.5293
Madigan, M. T., Martinko, J. M., & Parker, J. (2004). Brock Biología de los Microorganísmos, 10a Edición. Pearson Prentice Hall.
Mittenhuber, G., Sonomoto, K., Egert, M., & Friedrich, C. G. (1991). Identification of the DNA region responsible for sulfur-oxidizing ability of Thiosphaera pantotropha. J Bacteriol ., 7340–7344.
R,M y Bartha, R. (2002). Atlas Ecología microbiana y Microbiología ambiental. Pearson Prentice Hall.
Universidad Miguel Hernández de Elche. (2013). Producción de sulfhídrico. Prácticas de Microbiología.
Wodara, C., Kostka, S., Egert, M., Kelly, D. P., & Friedrich, C. G. (1994). Identification and sequence analysis of the soxB gene essential for sulfur oxidation of Paracoccus denitrificans GB17. J Bacteriol, 6188–6191.
Anexo


Aunque son posibles varios estados de oxidación (Brock, 2008) refiere que solo tres estados de oxidación, tienen importancia en la naturaleza.
-2 (sulfidrilo, R-SH y sulfuro HS-)
0 ( azufre elemental, S°).
+6 (Sulfato, SO42-).
Sulfuro de hidrógeno y reducción a sulfato

1 note
·
View note
Text
1 note
·
View note
Text


Una de mis pasiones es el diseño virtual, hice está imagen referente a nutrición, simulando cada espacio que ocupa un alimento en la posición de un numero de un reloj de manecillas y promoviendo la hidratación.
Una imagen es con fondo transparente y otra imagen es con fondo blanco.
1 note
·
View note
Text
Se realizó un problema hipotético, dónde para salvar especies en peligro de extinción se analizaron a través de muestras de distintos espermas seleccionados, el movimiento a través de distintos fotogramas seleccionados, con el fin de determinar su movimiento en espiral ascendente, ya que al estar en una muestra de portaobjetos para el microscopio su movimiento se limita en dos dimensiones. Por lo que comparto los cálculos usando la física que se realizó para predecir su movimiento en espiral ascendente y determinar la mejor muestra para salvar la especie.
1 note
·
View note
Text
EATING THE APPROACH FROM CONNECTOMICS
Azucena Jurado
Student of the Degree in Biotechnology, Faculty of Sciences
ABSTRACT
The activity of eating so far has three stages in which they are divided into: a) the cefalic phase b) the gastric phase and the intestinal phase; all three are regulated by neural activities. Likewise, there is documentation about the relationship between the brain areas activated at certain meal times. A database was made using the Braintome Atlas catalog, to evaluate the documented areas and the previously documented areas that are activated with three selected categories: a) Eating drinking, b) Perception gustation, c) Chewing / swallowing were selected. With these data, a matrix and a connectogram were elaborated, obtaining many of the documented connections, both theoretical and in the Braintome Atlas catalogue.
Introduction
The activity of eating so far has three stages in which it is divided, the cephalic phase, the gastric phase and the intestinal phase. During the cephalic phase of digestion, the smell. The initial sight, thought, or taste of food activates neuronal centers in the cerebral cortex, the hypothalamus, and the brain center.
The brainstem activates the facial (VII), glossopharyngeal (IX) and vagus (X) nerves. The fasial and glossopharyngeal nerves stimulate the secretion of saliva by the alivary glands, while the vagus nerve stimulates the gastric glands to produce gastric juice. The purpose of the cephalic phase of digestion is to prepare the mouth and stomach to receive the food that is to be ingested. The gastric phase and the intestinal phase have neural and hormonal regulation.
In the gastric phase, there are two receptors: the chemoreceptors, these are sensitive to the PH of the gastric quimo, and the gastric stretch receptors; When both receptors are activated, nerve impulses are sent to the submucosal plexus, where parasympathetic and enteric neurons are activated. These waves cause peristalsis and continue to stimulate the outflow of gastric acid. The intestinal phase is when food reaches the small intestine, there are inhibitory effects that slow the output of quimo from the stomach, this distension. It causes the enterogastric reflex, the receptors in charge send nerve impulses to the medulla oblongata where they inhibit parasympathetic stimulation and stimulate the sympathetic nerves of the stomach, thus inhibiting gastric motility and starting the contraction of the pyloric sphincter.
It is also important to analyze the functions that are registered in the areas of the brain in relation to eating. The hypothalamus is related to the control of appetite and satiety, there are receptor connections that send signals of hunger and satiety. Insular cortex one of its previously associated functions is the evaluation of the internal sensations of the body and is involved in the perception of taste and texture of food and is related to the feeling of satiety. With the background seen, a search was made in the Braintome Atlas catalogue; a conceptual map where the search for information and the information contained in the Braintom Atlas catalog were evaluated, and a connectagram was made with R studio based on the aforementioned relationships.
Method
1. It was previously documented about the functions of the brain area and connectomics.
2. The activity of eating was chosen to search for information in the Braintome Atlas database.
3. Through the Braintome Atlas, the areas of the brain that were activated when the activity was carried out were recorded.
4. Through the option of the connectogram, we began to analyze which of the selected areas were related. The categories of a) Eating drinking, b) Perception gustation, c) Chewing / swallowing were selected.
5. With the data collected, the list of active areas was recorded in a table and the matrix was prepared.
6. R Studio was used to make the relationship of brain functions.
7. Ran separate crypt to analyze trend measures.
Results
figure 1: conceptual map of feeding phases related to the activated areas recorded by the Braintome Atlas database

Braintome Atlas database. It can be observed that in the action of ¨Perception gustation¨ it belongs to the cephalic phase and the gastric phase and all the areas registered by Braintome Atlas (Subcortical Nuclei, Limbic Lobe, Insular Lobe, Frontal Lobe) are involved. In the gastric phase, it is observed that the frontal area does not act, there are only three areas that are activated: Subcortical Nuclei, Limbic Lobe, Insular Lobe.
In the action of "Chewing / swallowing" it is the one in which there is less registered in the Brainetome Atlas database, only the Frontal Lobe and Insular Lobe have been registered.
figure 2: Communities

This figure of nodes shows us that they form three communities, there are ways that separate three hemispheres, central, right and left hemisphere. It can be seen that in the extreme hemispheres there are few nodes.
figure 3: Adjacency

In the Braintome Atlas catalogue, the connectogram covers subareas in general, but does not cover subareas of subareas, so the matrix is 11 x 11.
Figure 4: Central measures a) Degree of centrality, b) closeness centrality, c) Betweenness centrality d) Hubs centrality, e) Authorities centrality, f) eigenvector Centrality






a) Degree of centrality: it is a measure of centrality that quantifies the frequency or the number of times that a node is between the geodesics or shortest paths of other actors. It can be seen that with the exception of node 6 and node 3, the other nodes interact with values of 8 and 9, with node 10 being the central node.
b) Closeness of centrality: is a measure that indicates the closeness centrality, it can be seen that both node 9 and 10 are the closest ones.
c) Betweenness centrality: betweenness is a measure of centrality that quantifies the frequency or number of times a node is found. You can see the nodes, 1, 4, 11, 7 as the most prominent.
d) Hubs centrality: is a measure that accounts for the quantity and quality of a node's connections.
e) Authorities centrality: it can be seen that only node 3 and 6 have no connection.
f) Eigenvector centrality: measures the eigenvector centrality, it can be seen that node 6 and 3 are distant from the central values.
Figure 5: Summary table and values

In the R Studio option, these windows are shown where they indicate the characteristic of the connectogram.
Discussion of results
(Harris, 2017) points out that the prefrontal cortex has been linked to decision making and planning. As well as the preparatory impulses in response to food. The associated pairs would be expected to be the same as those previously associated with the cephalic stage
It is known that in the gastric phase there are two receptors: the chemoreceptors that are sensitive to the quimo pH and the gastric stretch receptors; Both receptors, when activated, send nerve impulses to the submucosal plexus, where parasympathetic and enteric neurons are activated. These waves cause peristalsis and continue to stimulate gastric acid output, so it would be expected that all areas related to c) Chewing / swallowing and most of a) Eating drinking, are related in the database to the parasympathetic system.
The hypothalamus is related to the control of appetite and satiety, there are receptor connections that send signals of hunger and satiety, it is also expected that most or all of the selected pairs of category a) Eating drinking, are activated in this area.
The areas involved in the act of eating were analyzed. the three phases of digestion were known and associated. Programming in R was used to be able to associate a biological phenomenon.
Acknowledgment
A thank you to Doctor Ricardo Lara, for his effort in teaching us to use technology as part of the learning and study of Biophysics.
Bibliografía
Brainnetome Center, Institute of Automation. Connectogram [Internet]. BraintomeAtlas. 2014 [citado el 7 de junio de 2023]. Disponible en: https://atlas.brainnetome.org/
Tortota GJ, Derrickson B. Principios de Anatomía y Fisiología. Madrid, España: Editorial médica panamericana; 2015.
Annexes
1. Adjacency

2. Matrix


1 note
·
View note
Text
Vamos a desayunar enfrijoladas
1 note
·
View note
Text


Hoy comenzamos con el taller de Estrategia Nutricional para el Ciclismo
1 note
·
View note
Text
Quiero compartir con Ustedes unos dibujos en formato .png que realicé de la autoclave y proceso de esterilización de material de laboratorio.



#Autoclave#ajestrada#Azucena Jurado#Microbiología#prácticas de laboratorio#dibujo autoclave#esquema autoclave#ajestrada nutrición
1 note
·
View note
Text
1 note
·
View note
Text
Te comparto este recetario Navideño, deseando sea de tu apoyo en el proceso de mejorar tus hábitos.

1 note
·
View note




