#pyplot
Explore tagged Tumblr posts
Text

2025-06-20
Got through more of the opencv chapters.
#now its time to go outside and relax ~#studyblr#the new pycharm version finally supports jupyter notebooks hell yeah#but sadly pyplot is still not great in dark mode :( my eyes
0 notes
Text
pyplot is a type of maid who really doesn’t want you to talk to her but she’s good at her job
#i fear that api#i’m a shit programmer but like. it’s like they implemented the world’s most useful library using only side effects#to win a bet or something#python#codeblr#bathroom wall
3 notes
·
View notes
Note
Monthly reminder that Good god why did they make JavaScript like that? Which is it not object oriented? Why does it have objects anyways? Why are for each loops like that??? 1 == "true" returns true for some reason?
In other news, monthly reminder that you have a wonderful eye for outfits and it's always a pleasure to see you in nice outfit!
I hate hate HATE dynamically typed languages❗️‼️
I'm warming up to python a little bit just because numpy and pyplot is super convenient but I hate javascript so much it's unreal
12 notes
·
View notes
Text



RENDERING FRACTALS
Written in Python using numpy, pyplot, and numba for the jit compiler.
6 notes
·
View notes
Text
Contemporary Stroke Mortality in Florida by Jurisdiction
First we display boxplots of latitude and longitude reported within the chosen dataset for jurisdictions within Florida. Then we compare the extremes of these variables to a standard definition as follows (source: https://en.wikipedia.org/wiki/Florida). Latitude 24° 27' N to 31° 00' N Longitude 80° 02' W to 87° 38' W


We conclude that the extent of latitude and longitude of the entries in our dataset are congruent with a standard definition. Next we provide the script used to generate our findings:
set current working directory
from os import chdir, getcwd wd=getcwd() chdir(wd)
import libraries
import pandas import numpy
read in data
data = pandas.read_csv('C:/Users/jrhud/OneDrive/Desktop/DataMgmtVizProject/Stroke_Mortality_ Data_Among_US_Adults__35___by_State_Territory_and_County___2019-2021.csv')
low_memory does not work like it should
check for accurate result of data import
print(len(data)) print(len(data.columns))
subset to florida
dataFl = data[data['LocationAbbr']=='FL']
subset Florida to columns of interest
subFl = dataFl[['Data_Value', 'Y_lat', 'X_lon']]
check for accurate result of data subset
print(len(subFl)) print(len(subFl.columns))
import library for boxplot
from matplotlib import pyplot as plt
create boxplots for DataValue, Y_lat, and Xz-lonsubFl
subFl['Data_Value'].plot(kind = 'box', title = "Stroke Mortality by Jurisdiction within Florida") plt.show() subFl['Y_lat'].plot(kind = 'box', title = "Latitude of Jurisdictions within Florida") plt.show() subFl['X_lon'].plot(kind = 'box', title = "Longitude of Jurisdictions within Florida") plt.show() Finally, we report our boxplot of contemporary stroke mortality by jurisdiction within the state of Florida. Further analysis will be reported as the assignment is completed.

0 notes
Text
Productivity challenge #1
Completed my vacation work in one go hehe yay!
+Read the introduction to Python Pandas and PyPlot and practiced MySQL queries


1 note
·
View note
Text
What libraries do data scientists use to plot data in Python?
Hi,
When it comes to visualizing data in Python, data scientists have a suite of powerful libraries at their disposal. Each library has unique features that cater to different types of data visualization needs:
Matplotlib: Often considered the foundational plotting library in Python, Matplotlib offers a wide range of plotting capabilities. Its pyplot module is particularly popular for creating basic plots such as line charts, scatter plots, bar graphs, and histograms. It provides extensive customization options to adjust every aspect of a plot.
Seaborn: Built on top of Matplotlib, Seaborn enhances the aesthetics of plots and simplifies the creation of complex visualizations. It excels in generating attractive statistical graphics such as heatmaps, violin plots, and pair plots, making it a favorite for exploring data distributions and relationships.
Pandas: While primarily known for its data manipulation capabilities, Pandas integrates seamlessly with Matplotlib to offer quick and easy plotting options. DataFrames in Pandas come with built-in methods to generate basic plots, such as line plots, histograms, and bar plots, directly from the data.
Plotly: This library is geared towards interactive plots. Plotly allows users to create complex interactive charts that can be embedded in web applications. It supports a wide range of chart types and interactive features like zooming and hovering, making it ideal for presentations and dashboards.
Altair: Known for its concise syntax and declarative approach, Altair is used to create interactive visualizations with minimal code. It’s especially good for handling large datasets and generating charts like bar charts, scatter plots, and line plots in a clear and straightforward manner.
Bokeh: Bokeh specializes in creating interactive and real-time streaming plots. It’s particularly useful for building interactive dashboards and integrating plots into web applications. Bokeh supports various types of plots and offers flexibility in customizing interactions.

Each of these libraries has its strengths, and data scientists often use them in combination to leverage their unique features and capabilities, ensuring effective and insightful data visualization.
Drop the message to learn more!!
0 notes
Text
Task
This week’s assignment involves decision trees, and more specifically, classification trees. Decision trees are predictive models that allow for a data driven exploration of nonlinear relationships and interactions among many explanatory variables in predicting a response or target variable. When the response variable is categorical (two levels), the model is a called a classification tree. Explanatory variables can be either quantitative, categorical or both. Decision trees create segmentations or subgroups in the data, by applying a series of simple rules or criteria over and over again which choose variable constellations that best predict the response (i.e. target) variable.
Run a Classification Tree.
You will need to perform a decision tree analysis to test nonlinear relationships among a series of explanatory variables and a binary, categorical response variable.
Data
Features are computed from a digitized image of a fine needle aspirate (FNA) of a breast mass [K. P. Bennett and O. L. Mangasarian: "Robust Linear Programming Discrimination of Two Linearly Inseparable Sets", Optimization Methods and Software 1, 1992, 23-34].
Dataset can be found at UCI Machine Learning Repository
In this Assignment the Decision tree has been applied to classification of breast cancer detection.
Attribute Information:
id - ID number
diagnosis (M = malignant, B = benign)
3-32 extra features
Ten real-valued features are computed for each cell nucleus: a) radius (mean of distances from center to points on the perimeter) b) texture (standard deviation of gray-scale values) c) perimeter d) area e) smoothness (local variation in radius lengths) f) compactness (perimeter^2 / area - 1.0) g) concavity (severity of concave portions of the contour) h) concave points (number of concave portions of the contour) i) symmetry j) fractal dimension ("coastline approximation" - 1)
All feature values are recoded with four significant digits. Missing attribute values: none Class distribution: 357 benign, 212 malignant
Results
Generated decision tree can be found below:
In [17]:img
Out[17]:
Decision tree analysis was performed to test nonlinear relationships among a series of explanatory variables and a binary, categorical response variable (breast cancer diagnosis: malignant or benign).
The dataset was splitted into train and test samples in ratio 70\30.
After fitting the classifier the key metrics were calculated - confusion matrix and accuracy = 0.924. This is a good result for a model trained on a small dataset.
From decision tree we can observe:
The malignant tumor is tend to have much more visible affected areas, texture and concave points, while the benign's characteristics are significantly lower.
The most important features are:
concave points_worst = 0.707688
area_worst = 0.114771
concave points_mean = 0.034234
fractal_dimension_se = 0.026301
texture_worst = 0.026300
area_se = 0.025201
concavity_se = 0.024540
texture_mean = 0.023671
perimeter_mean = 0.010415
concavity_mean = 0.006880
Code
In [1]:import pandas as pd import numpy as np from sklearn.metrics import*from sklearn.model_selection import train_test_split from sklearn.tree import DecisionTreeClassifier from sklearn import tree from io import StringIO from IPython.display import Image import pydotplus from sklearn.manifold import TSNE from matplotlib import pyplot as plt %matplotlib inline rnd_state = 23468
Load data
In [2]:data = pd.read_csv('Data/breast_cancer.csv') data.info() <class 'pandas.core.frame.DataFrame'> RangeIndex: 569 entries, 0 to 568 Data columns (total 33 columns): id 569 non-null int64 diagnosis 569 non-null object radius_mean 569 non-null float64 texture_mean 569 non-null float64 perimeter_mean 569 non-null float64 area_mean 569 non-null float64 smoothness_mean 569 non-null float64 compactness_mean 569 non-null float64 concavity_mean 569 non-null float64 concave points_mean 569 non-null float64 symmetry_mean 569 non-null float64 fractal_dimension_mean 569 non-null float64 radius_se 569 non-null float64 texture_se 569 non-null float64 perimeter_se 569 non-null float64 area_se 569 non-null float64 smoothness_se 569 non-null float64 compactness_se 569 non-null float64 concavity_se 569 non-null float64 concave points_se 569 non-null float64 symmetry_se 569 non-null float64 fractal_dimension_se 569 non-null float64 radius_worst 569 non-null float64 texture_worst 569 non-null float64 perimeter_worst 569 non-null float64 area_worst 569 non-null float64 smoothness_worst 569 non-null float64 compactness_worst 569 non-null float64 concavity_worst 569 non-null float64 concave points_worst 569 non-null float64 symmetry_worst 569 non-null float64 fractal_dimension_worst 569 non-null float64 Unnamed: 32 0 non-null float64 dtypes: float64(31), int64(1), object(1) memory usage: 146.8+ KB
In the output above there is an empty column 'Unnamed: 32', so next it should be dropped.
In [3]:data.drop('Unnamed: 32', axis=1, inplace=True) data.diagnosis = np.where(data.diagnosis=='M', 1, 0) # Decode diagnosis into binary data.describe()
Out[3]:iddiagnosisradius_meantexture_meanperimeter_meanarea_meansmoothness_meancompactness_meanconcavity_meanconcave points_mean...radius_worsttexture_worstperimeter_worstarea_worstsmoothness_worstcompactness_worstconcavity_worstconcave points_worstsymmetry_worstfractal_dimension_worstcount5.690000e+02569.000000569.000000569.000000569.000000569.000000569.000000569.000000569.000000569.000000...569.000000569.000000569.000000569.000000569.000000569.000000569.000000569.000000569.000000569.000000mean3.037183e+070.37258314.12729219.28964991.969033654.8891040.0963600.1043410.0887990.048919...16.26919025.677223107.261213880.5831280.1323690.2542650.2721880.1146060.2900760.083946std1.250206e+080.4839183.5240494.30103624.298981351.9141290.0140640.0528130.0797200.038803...4.8332426.14625833.602542569.3569930.0228320.1573360.2086240.0657320.0618670.018061min8.670000e+030.0000006.9810009.71000043.790000143.5000000.0526300.0193800.0000000.000000...7.93000012.02000050.410000185.2000000.0711700.0272900.0000000.0000000.1565000.05504025%8.692180e+050.00000011.70000016.17000075.170000420.3000000.0863700.0649200.0295600.020310...13.01000021.08000084.110000515.3000000.1166000.1472000.1145000.0649300.2504000.07146050%9.060240e+050.00000013.37000018.84000086.240000551.1000000.0958700.0926300.0615400.033500...14.97000025.41000097.660000686.5000000.1313000.2119000.2267000.0999300.2822000.08004075%8.813129e+061.00000015.78000021.800000104.100000782.7000000.1053000.1304000.1307000.074000...18.79000029.720000125.4000001084.0000000.1460000.3391000.3829000.1614000.3179000.092080max9.113205e+081.00000028.11000039.280000188.5000002501.0000000.1634000.3454000.4268000.201200...36.04000049.540000251.2000004254.0000000.2226001.0580001.2520000.2910000.6638000.207500
8 rows × 32 columns
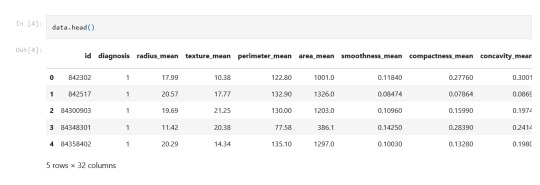
In [4]:data.head()
Out[4]:iddiagnosisradius_meantexture_meanperimeter_meanarea_meansmoothness_meancompactness_meanconcavity_meanconcave points_mean...radius_worsttexture_worstperimeter_worstarea_worstsmoothness_worstcompactness_worstconcavity_worstconcave points_worstsymmetry_worstfractal_dimension_worst0842302117.9910.38122.801001.00.118400.277600.30010.14710...25.3817.33184.602019.00.16220.66560.71190.26540.46010.118901842517120.5717.77132.901326.00.084740.078640.08690.07017...24.9923.41158.801956.00.12380.18660.24160.18600.27500.08902284300903119.6921.25130.001203.00.109600.159900.19740.12790...23.5725.53152.501709.00.14440.42450.45040.24300.36130.08758384348301111.4220.3877.58386.10.142500.283900.24140.10520...14.9126.5098.87567.70.20980.86630.68690.25750.66380.17300484358402120.2914.34135.101297.00.100300.132800.19800.10430...22.5416.67152.201575.00.13740.20500.40000.16250.23640.07678
5 rows × 32 columns
Plots
For visualization purposes, the number of dimensions was reduced to two by applying t-SNE method. The plot illustrates that our classes are not clearly divided into two parts, so the nonlinear methods (like Decision tree) may solve this problem.
In [15]:model = TSNE(random_state=rnd_state, n_components=2) representation = model.fit_transform(data.iloc[:, 2:])
In [16]:plt.scatter(representation[:, 0], representation[:, 1], c=data.diagnosis, alpha=0.5, cmap=plt.cm.get_cmap('Set1', 2)) plt.colorbar(ticks=range(2));
Decision tree
In [6]:predictors = data.iloc[:, 2:] target = data.diagnosis
To train a Decision tree the dataset was splitted into train and test samples in proportion 70/30.
In [7]:(predictors_train, predictors_test, target_train, target_test) = train_test_split(predictors, target, test_size = .3, random_state = rnd_state)
In [8]:print('predictors_train:', predictors_train.shape) print('predictors_test:', predictors_test.shape) print('target_train:', target_train.shape) print('target_test:', target_test.shape) predictors_train: (398, 30) predictors_test: (171, 30) target_train: (398,) target_test: (171,)
In [9]:print(np.sum(target_train==0)) print(np.sum(target_train==1)) 253 145
Our train sample is quite balanced, so there is no need in balancing it.
In [10]:classifier = DecisionTreeClassifier(random_state = rnd_state).fit(predictors_train, target_train)
In [11]:prediction = classifier.predict(predictors_test)
In [12]:print('Confusion matrix:\n', pd.crosstab(target_test, prediction, colnames=['Actual'], rownames=['Predicted'], margins=True)) print('\nAccuracy: ', accuracy_score(target_test, prediction)) Confusion matrix: Actual 0 1 All Predicted 0 96 8 104 1 5 62 67 All 101 70 171 Accuracy: 0.9239766081871345
In [13]:out = StringIO() tree.export_graphviz(classifier, out_file = out, feature_names = predictors_train.columns.values, proportion =True, filled =True) graph = pydotplus.graph_from_dot_data(out.getvalue()) img = Image(data = graph.create_png()) with open('output.png', 'wb') as f: f.write(img.data)
In [14]:feature_importance = pd.Series(classifier.feature_importances_, index=data.columns.values[2:]).sort_values(ascending=False) feature_importance
Out[14]:concave points_worst 0.707688 area_worst 0.114771 concave points_mean 0.034234 fractal_dimension_se 0.026301 texture_worst 0.026300 area_se 0.025201 concavity_se 0.024540 texture_mean 0.023671 perimeter_mean 0.010415 concavity_mean 0.006880 fractal_dimension_worst 0.000000 fractal_dimension_mean 0.000000 symmetry_mean 0.000000 compactness_mean 0.000000 texture_se 0.000000 smoothness_mean 0.000000 area_mean 0.000000 radius_se 0.000000 smoothness_se 0.000000 perimeter_se 0.000000 symmetry_worst 0.000000 compactness_se 0.000000 concave points_se 0.000000 symmetry_se 0.000000 radius_worst 0.000000 perimeter_worst 0.000000 smoothness_worst 0.000000 compactness_worst 0.000000 concavity_worst 0.000000 radius_mean 0.000000 dtype: float64
0 notes
Text
อันดับทีมชาติฟุตบอลที่ไหนเป็นเว็บไซต์ที่น่าเชื่อถือสำหรับการพนัน?
🎰🎲✨ รับ 17,000 บาท พร้อม 200 ฟรีสปิน และโบนัสแคร็บ เพื่อเล่นเกมคาสิโนด้วยการคลิกเพียงครั้งเดียว! ✨🎲🎰
อันดับทีมชาติฟุตบอลที่ไหนเป็นเว็บไซต์ที่น่าเชื่อถือสำหรับการพนัน?
พบกับเว็บไซต์พนันฟุตบอลที่มีคุณภาพและน่าเชื่อถือได้บนโลกออนไลน์จำนวนมาก การเลือกเว็บไซต์ในการแทงบอลออนไลน์อาจทำให้คุณรู้สึกสับสนมาก เนื่องจากมีหลายที่ให้บริการ ดังนั้นจึงเป็นสิ่งสำคัญที่ให้ความสำคัญกับเกมคาสิโนออนไลน์ประเภทนี้ เมื่อคุณเลือกเว็บไซต์ที่ได้รับการรับรองและนิสิตถุให้ชื่อเสียง ความไว้วางใจในการเดิมพันแล้วสามารถเพิกเกมทำเงินได้จริงๆ ไม่ว่าจะเป็นการแทงบอล บาคาร่า หรือไพ่โป๊กเกอร์
เว็บไซต์ที่ดีมักมีระบบความเสถียรและรักษาความลับของข้อมูลส่วนบุคคลของผู้เล่นอย่างเข้มงวด นอกจากนี้ มีระบบการเงินที่ปลอดภัยและมีความรับผิดชอบอย่างสูง อย่างไรก็ตาม จำเป็นต้องระมัดระวังกับการเดิมพันออนไลน์ และไม่ควรใช้ชีวิตส่วนตัวเพื่อเดิมพัน
หากต้องการเลือกเว็บไซต์พนันฟุตบอลที่ดี ควรค้นคว้าข้อมูลและอ่านรีวิวจากผู้เล่นที่มีประสบการณ์ โดยเลือกเว็บไซต์ที่มีข้อดีในการให้บริการ ทำให้คุณรับประสบการณ์เดิมพันที่ดีและปลอดภัย อย่างเช่นเพจบอลทั้งแพ้และชนะ สูง/ต่ำ และโอเวอร์/ยอร์
โดยรวม การเลือกเว็บไซต์พนันฟุตบอลที่เป็นผู้นำในวงการอย่างดีจะมีผลต่อประสิทธิภาพและประสิทธิผลของการเดิมพันของคุณในระยะยาว
ทีมชาติฟุตบอลใหนที่น่าเชื่อถือกันมากที่สุด? การเลือกทีมชาติฟุตบอลที่น่าเชื่อถือไม่ใช่เรื่องง่าย เนื่องจากมีทีมที่มีประสบการณ์และความสามารถมากมายในการแข่งขัน อย่างไรก็ตาม มีหลายปัจจัยที่ต้องพิจารณาเมื่อเลือกทีมที่น่าเชื่อถืออย่างแท้จริง
หนึ่งในทีมชาติฟุตบอลที่น่าเชื่อถือที่สุดในปัจจุบันคือทีมชาติบราซิล ทีมชาตินี้มีประวัติการแข่งขันที่ยาวนานและมีผลงานที่ยิ่งใหญ่ในฟุตบอลโลก ไม่ว่าจะเป็นการคว้าแชมป์หรือการแข่งขันในระดับสูง ทีมชาติบราซิตอบริจุดเริ่มต้นของฟุตบอลทั้งโลก
อีกทีมหนึ่งที่น่าเชื่อถือไม่แพ้ไม่ร้อนจากนั้นคือทีมชาติเยอรมนี ทีมชาตินี้มีความสามารถในการเล่นที่สูงและมีความเชี่ยวชาญในการจัดการเกมและสมรรถที่ในการทำประตู ทีมชาติเยอรมนีเป็นแกรนด์สแลมของโลกที่หลายคนเชื่อว่าสามารถเอาชนะใครก็ได้ในแข่งขันใดๆ
ไม่ว่าจะเลือกทีมชาติใด ทีมชาติที่น่าเชื่อถือควรมีความองค์รวมและความพร้อมที่สูง เพื่อที่จะทำให้ผลลัพธ์ที่ดีในการแข่งขัน ทางานขอรีบขอใจๆเป็นทีมชาติเชื่อถือถือทะลายได้แปลงเปลี่ยนสภาพการแข่งขันให้ดีขึ้น ให้เล่นเพื่อเป็นสีเอกสุดความสามารถแล้วลุขียุงัย่างแจ้งาง้อ.pyplot
ทีมชาติฟุตบอลเป็นหนึ่งในกลุ่มทีมที่มีความน่าสนใจในการพนันเป็นอย่างมาก มีหลายปัจจัยที่ทำให้ทีมชาติบางทีน่าสนใจกว่า��ีมอื่น ๆ ในการเดิมพัน เช่น มีการชนะในการแข่งขันมากมายหรือมีนักเตะชื่อดังที่น่าสนใจ
ทีมชาติบางชุมมักมีประวัติศาสตร์ที่แข็งแกร่งและความเป็นผู้นำในวงการเรกู๊และที่ดีเยี่ยม ซึ่งทำให้กระนั้นทีมเหล่านี้มักมีการพนันได้รับความนิยม ตัวอย่างเช่น ทีมชาติบราซิลที่มีประวัติศาสตร์แข็งแกร่งในวงการบ๊อล เเละได้ชนะในรายการการแข่งขันระดับโลกระดับสูง ซึ่งส่งผลให้มีการพนันในทีมดังนี้มีตลาดข้องเสียภาพ และน่าสนใจเสมอ
นอกจากนี้ การสร้างความพึงพอใจกลุ่มผู้เล่นที่ทำการพนันกับทีมชาติ สำคัญอย่างมาก ผ่านการศึกษาผลงานระดับโลกและการวิเศษจากนักกีฬาดีเด่น เช่น ซาลาร์ คูด ของทีมชาติสเปน นักยิงและนักยอดชั้นเลิศ ทำให้ทีมชาตินี้มีการติดประวติยีท่ที่สู่ที่ติดต่า
จากข้อมูลข้างต้น ทีมชาติที่เหมาะสมสำหรับพนันคือทีมที่แข็งแกร่ขการทำยอย มีประวติย่าสสตร์ทีทุเดีท่ดีเยี่ยเทาเทนีี่่ใ堂่าี่ย่าうํี่主ยบอยด่ายน่าสนใจ ปรอใยมเ而ืีี๋ี原ใ่็บย少่ายท่至。
ในโลกของการพนันฟุตบอลออนไลน์, การเข้าถึงแหล่งข้อมูลที่เชื่อถือได้เป็นสิ่งจำเป็นที่ช่วยให้นักพนันมีโอกาสชนะเดิมพันได้มากขึ้น แม้ว่ามีหลายแหล่งข้อมูลในเว็บไซต์ที่เสฉองว่าช่วยเสริมความเชื่อมั่นของนักพนัน แต่ไม่ทุกแหล่งข้อมูลสามารถเชื่อถือได้ การดำเนินการค้นหาข้อมูลจากแหล่งที่เชื่อถือได้จึงเป็นสิ่งสำคัญอย่างยิ่ง
หนึ่งในแหล่งข้อมูลที่นักพนันฟุตบอลควรทราบคือเว็บไซต์ที่มีบทวิเคราะห์การแข่งขัน ข่าวสารสําคัญ และสถิติต่างๆ เพื่อช่วยในการตัดสินใจในการวางเดิมพันได้อย่างมั่นใจ และเพื่อป้องกันการเสียเงินโดยเปล่าประโยชน์
นอกจากนั้น, การเข้าถึงแหล่งข้อมูลเพื่อการพนันฟุตบอลยังสามารถช่วยให้นักพนันสามารถวิเคราะห์สถานการณ์ทีมที่แข็งแกร่งและเบอร์หรือมีผลงานที่ดีในการแข่งขัน เพื่อช่วยให้เสมอฟุตบอลที่สามารถประยุกต์หรือวางเดิมพันได้อย่างมั่นใจ
ด้วยข้อมูลที่สมบรูณ์และแม่นยำ, นักพนันฟุตบอลจะมีโอกาสในการชนะเดิมพันมากขึ้น การค้นหาและเรียนรู้จากแหล่งข้อมูลที่เชื่อถือได้จึงเป็นสิ่งสำคัญสําหรับนักเสมอฟุตบอลทุกคนที่ต้องการสร้างรายได้จากการเดิมพันถูกต้องและประสบความสำเร็จในวงการนี้
ทำการพนันออนไลน์อาจเป็นเรื่องที่ยากที่จะหาเว็บไซต์ที่มีความน่าเชื่อถือ แต่มีเว็บไซต์ค่อนข้างเป็นที่รู้จักในวงการนี้ ซึ่งจะช่วยให้ผู้เล่นสามารถเข้าเล่นมาและมั่นใจได้ ดังนี้คือรายการของ 5 เว็บไซต์พนันที่มีความน่าเชื่อถือ:
888casino - เป็นเว็บไซต์พนันที่มีชื่อเสียงอย่างเชื่อถือได้และมีการให้บริการความสนุกสนานแก่ผู้เล่นมานานกว่า 20 ปี
Betway - เว็บไซต์นี้เน้นการให้บริการทางด้านการพนันออกมาอย่างม��ออาชีพ และมีความน่าเชื่อถือสูง
LeoVegas - มีเกมส์คาสิโนและสล็อตที่หลากหลาย และมีความปลอดภัยสูง
22BET - เว็บไซต์ที่มีการให้บริการเกมส์คาสิโนและพนันออนไลน์ที่น่าเชื่อถือ
Royal Panda - เป็นเว็บไซต์ที่มีความน่าเชื่อถือและมีเกมส์คาสิโนที่หลากหลายให้ผู้เล่นได้เลือกเล่น
การเลือกเว็บไซต์พนันที่มีความน่าเชื่อถือเป็นสิ่งสำคัญอย่างยิ่ง เพื่อป้องกันการเสียเงินอย่างไม่จำเป็น ผู้เล่นควรทำการค้นคว้าข้อมูลและรีวิวจากผู้เล่นคนอื่นเพื่อทำให้ตัดสินใจได้อย่างมั่นใจและเห็นแนวโน้มของเว็บไซต์ และอย่าลืมเล่นให้มีความสนุกสนาน และดูแลตัวเองเป็นสัดส่วนในการพนันออนไลน์ได้อย่างดี
0 notes
Text
Running a Classification Tree
This week’s assignment involves decision trees, and more specifically, classification trees. Decision trees are predictive models that allow for a data driven exploration of nonlinear relationships and interactions among many explanatory variables in predicting a response or target variable. When the response variable is categorical (two levels), the model is a called a classification tree. Explanatory variables can be either quantitative, categorical or both. Decision trees create segmentation or subgroups in the data, by applying a series of simple rules or criteria over and over again which choose variable constellations that best predict the response (i.e. target) variable.
Run a Classification Tree.
Data
Features are computed from a digitized image of a fine needle aspirate (FNA) of a breast mass [K. P. Bennett and O. L. Mangasarian: "Robust Linear Programming Discrimination of Two Linearly Inseparable Sets", Optimization Methods and Software 1, 1992, 23-34].
In this Assignment the Decision tree has been applied to classification of breast cancer detection.
Attribute Information:
id - ID number
diagnosis (M = malignant, B = benign)
3-32 extra features
Ten real-valued features are computed for each cell nucleus: a) radius (mean of distances from center to points on the perimeter) b) texture (standard deviation of gray-scale values) c) perimeter d) area e) smoothness (local variation in radius lengths) f) compactness (perimeter^2 / area - 1.0) g) concavity (severity of concave portions of the contour) h) concave points (number of concave portions of the contour) i) symmetry j) fractal dimension ("coastline approximation" - 1)
All feature values are recorded with four significant digits. Missing attribute values: none Class distribution: 357 benign, 212 malignant
Results
Generated decision tree can be found below:
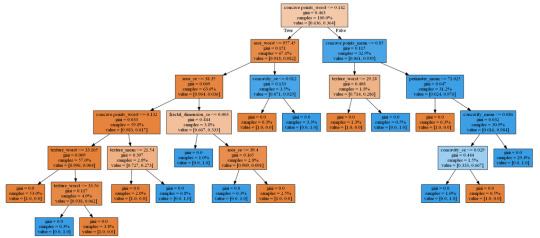
Decision tree analysis was performed to test nonlinear relationships among a series of explanatory variables and a binary, categorical response variable (breast cancer diagnosis: malignant or benign).
The dataset was splitted into train and test samples in ratio 70\30.
After fitting the classifier the key metrics were calculated - confusion matrix and accuracy = 0.924. This is a good result for a model trained on a small dataset.
From decision tree we can observe:
The malignant tumor is tend to have much more visible affected areas, texture and concave points, while the benign's characteristics are significantly lower.
The most important features are:
concave points_worst = 0.707688
area_worst = 0.114771
concave points_mean = 0.034234
fractal_dimension_se = 0.026301
texture_worst = 0.026300
area_se = 0.025201
concavity_se = 0.024540
texture_mean = 0.023671
perimeter_mean = 0.010415
concavity_mean = 0.006880
Code
import pandas as pd
import numpy as np
from sklearn.metrics import * from sklearn.model_selection
import train_test_split from sklearn.tree
import DecisionTreeClassifier
from sklearn import tree
from io import StringIO
from IPython.display import Image
import pydotplus
from sklearn.manifold import TSNE
from matplotlib import pyplot as plt
%matplotlib inline
rnd_state = 23467
Load data
data = pd.read_csv('Data/breast_cancer.csv')
data.info()
Output:
<class 'pandas.core.frame.DataFrame'> RangeIndex: 569 entries, 0 to 568 Data columns (total 33 columns): id 569 non-null int64 diagnosis 569 non-null object radius_mean 569 non-null float64 texture_mean 569 non-null float64 perimeter_mean 569 non-null float64 area_mean 569 non-null float64 smoothness_mean 569 non-null float64 compactness_mean 569 non-null float64 concavity_mean 569 non-null float64 concave points_mean 569 non-null float64 symmetry_mean 569 non-null float64 fractal_dimension_mean 569 non-null float64 radius_se 569 non-null float64 texture_se 569 non-null float64 perimeter_se 569 non-null float64 area_se 569 non-null float64 smoothness_se 569 non-null float64 compactness_se 569 non-null float64 concavity_se 569 non-null float64 concave points_se 569 non-null float64 symmetry_se 569 non-null float64 fractal_dimension_se 569 non-null float64 radius_worst 569 non-null float64 texture_worst 569 non-null float64 perimeter_worst 569 non-null float64 area_worst 569 non-null float64 smoothness_worst 569 non-null float64 compactness_worst 569 non-null float64 concavity_worst 569 non-null float64 concave points_worst 569 non-null float64 symmetry_worst 569 non-null float64 fractal_dimension_worst 569 non-null float64 Unnamed: 32 0 non-null float64 dtypes: float64(31), int64(1), object(1) memory usage: 146.8+ KB
In the output above there is an empty column 'Unnamed: 32', so next it should be dropped.

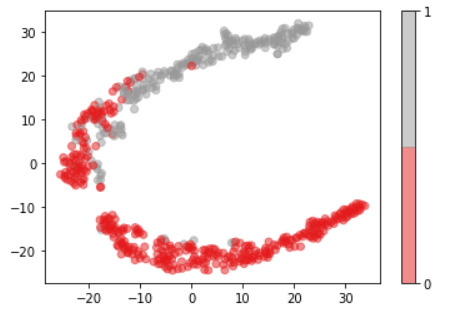
Plots
For visualization purposes, the number of dimensions was reduced to two by applying t-SNE method. The plot illustrates that our classes are not clearly divided into two parts, so the nonlinear methods (like Decision tree) may solve this problem.
model = TSNE(random_state=rnd_state, n_components=2) representation = model.fit_transform(data.iloc[:, 2:])
plt.scatter(representation[:, 0], representation[:, 1], c=data.diagnosis, alpha=0.5, cmap=plt.cm.get_cmap('Set1', 2)) plt.colorbar(ticks=range(2));
Decision tree
predictors = data.iloc[:, 2:]
target = data.diagnosis
To train a Decision tree the dataset was splitted into train and test samples in proportion 70/30.
(predictors_train, predictors_test, target_train, target_test) = train_test_split(predictors, target, test_size = .3, random_state = rnd_state)
print('predictors_train:', predictors_train.shape) print('predictors_test:', predictors_test.shape) print('target_train:', target_train.shape) print('target_test:', target_test.shape)
Output:
predictors_train: (398, 30)
predictors_test: (171, 30)
target_train: (398,)
target_test: (171,)
print(np.sum(target_train==0))
print(np.sum(target_train==1))
Output:
253
145
Our train sample is quite balanced, so there is no need in balancing it.
classifier = DecisionTreeClassifier(random_state = rnd_state).fit(predictors_train, target_train)
prediction = classifier.predict(predictors_test)
print('Confusion matrix:\n', pd.crosstab(target_test, prediction, colnames=['Actual'], rownames=['Predicted'], margins=True)) print('\nAccuracy: ', accuracy_score(target_test, prediction))

out = StringIO() tree.export_graphviz(classifier, out_file = out, feature_names = predictors_train.columns.values, proportion = True, filled = True) graph = pydotplus.graph_from_dot_data(out.getvalue()) img = Image(data = graph.create_png()) with open('output.png', 'wb') as f: f.write(img.data)
feature_importance = pd.Series(classifier.feature_importances_, index=data.columns.values[2:]).sort_values(ascending=False) feature_importance
concave points_worst 0.705688 area_worst 0.214871 concave points_mean 0.034234 fractal_dimension_se 0.028301 texture_worst 0.026300 area_se 0.025201 concavity_se 0.024540 texture_mean 0.023671 perimeter_mean 0.010415 concavity_mean 0.006880 fractal_dimension_worst 0.000000 fractal_dimension_mean 0.000000 symmetry_mean 0.000002 compactness_mean 0.005000 texture_se 0.000000 smoothness_mean 0.000000 area_mean 0.000000 radius_se 0.000000 smoothness_se 0.000000 perimeter_se 0.000001 symmetry_worst 0.000000 compactness_se 0.000000 concave points_se 0.000000 symmetry_se 0.000000 radius_worst 0.000000 perimeter_worst 0.000000 smoothness_worst 0.000000 compactness_worst 0.000002 concavity_worst 0.000000 radius_mean 0.000000 dtype: float64
#decision tree
1 note
·
View note
Text
also as of me writing this, this page tells you "you should use the explicit interface, see this other page for details on what we mean by this". and then, as far as I can tell, explains how to perform this procedure using the implicit interface they just advised us not to use
matplotlib what are you doing
*argsint, (int, int, index), or SubplotSpec, default: (1, 1, 1) The position of the subplot described by one of Three integers (nrows, ncols, index). The subplot will take the index position on a grid with nrows rows and ncols columns. index starts at 1 in the upper left corner and increases to the right. index can also be a two-tuple specifying the (first, last) indices (1-based, and including last) of the subplot, e.g., fig.add_subplot(3, 1, (1, 2)) makes a subplot that spans the upper 2/3 of the figure.
listen. i have my own opinions on 0-based indexing vs 1-based indexing. a language's developers are free to choose their own standard. personally i prefer 1-based, for reasons i'll get into later
but mixing indexing conventions like this is unconscionable. you can't do this! you can't be introducing 1-based indexing into a new context in a 0-based language!
1 note
·
View note
Photo

A journey of a thousand miles begins with but a single step. - Li Er . After months of inactivity, I decided to get off my ass and walk at least 10k steps a day. This is where we are after 50 days of that decision. Tried doing it for the past 2 years, failing every time. Reaching the 50-day milestone was pleasant if nothing else. So decided to document it. . The visualization is created in Python using pandas, matplotlib, and pyplot modules. . If interested, DM to collaborate on projects.
0 notes
Photo

Python has the ability to create graphs by using the matplotlib library. It has numerous packages and functions which generate a wide variety of graphs and plots. Check our Info : www.incegna.com Reg Link for Programs : http://www.incegna.com/contact-us Follow us on Facebook : www.facebook.com/INCEGNA/? Follow us on Instagram : https://www.instagram.com/_incegna/ For Queries : [email protected] #python,#frpahplotting,#matplotlib,#pandas,#numpy,#pyplot,#MATLAB,#visualizations,#linegraph,#subplot,#Contourplot,#API,#objectoriented,#oopsinpython,#deeplearning,#machinelearning https://www.instagram.com/p/B-MXeuQgYJ2/?igshid=7xkjex94f883
#python#frpahplotting#matplotlib#pandas#numpy#pyplot#matlab#visualizations#linegraph#subplot#contourplot#api#objectoriented#oopsinpython#deeplearning#machinelearning
0 notes
Text
*facing Pyplot*
So here's the deal. I could use you, or Matplotlib over there. The only thing you have going for you is that you make relatively easy dropdown menus. However, that's a thing you do in Dash, and I'm not sure if I give you to my professor he'll be able to run it.
Matplotlib, you're not great. But I do know that you're able to draw rectangles easily on an axis. So that's honestly a five-star review right there.
Bruh, I'm a geologist. I've taken .5 coding classes. Idk why I thought drawing squares on a graph in Python would be easy.
11 notes
·
View notes
Text
matplotlib – pyplot scatter plot marker size
This can be a somewhat confusing way of defining the size but you are basically specifying the area of the marker. This means, to double the width (or height) of the marker you need to increase s by a factor of 4. [because A = WH => (2W)(2H)=4A]
There is a reason, however, that the size of markers is defined in this way. Because of the scaling of area as the square of width, doubling the width actually appears to increase the size by more than a factor 2 (in fact it increases it by a factor of 4). To see this consider the following two examples and the output they produce.
0 notes